아연 집게


아연 집게(영어: zinc finger)는 단백질의 접힌 부분을 안정시키기 위해 하나 이상의 아연 이온(Zn2+)이 존재하는 작은 구조 단백질 모티브다. 원래 아프리카발톱개구리의 전사인자 IIIA로부터 가정된 구조의 집게같은 외관을 설명하기 위해 만들어진 아연 집게는 매우 다양한 단백질 구조를 포괄하게 되었다.[1] 아프리카발톱개구리의 TFIIIA는 원래 아연을 함유하고 기능을 위해 금속을 필요로 하는 것으로 1983년에 입증되었다. 유전자 조절 단백질의 아연이 필요하다는 것이 처음 보고되었다.[2][3] 다중 도메인 단백질에서 금속 결합 도메인으로 나타나는 경우가 많다.
아연 집게를 포함하는 단백질(아연 집게 단백질)은 여러 가지 다른 구조적 계열로 분류된다. 격자 모양이나 베타 헤어핀 구조와 같이 명확하게 정의되어 있는 많은 다른 2차 구조와는 달리, 각각 독특한 3차원 구조를 가진 아연 집게의 종류가 있다. 특정 아연 집게 단백질의 등급은 이 3차원 구조로 결정되지만, 아연 이온을 조정하는 단백질의 1차 구조나 리간드의 동일성에 근거하여도 인정될 수 있다. 그러나 이러한 단백질의 많은 다양성에도 불구하고, 대다수의 단백질은 일반적으로 DNA, RNA, 단백질 또는 기타 작고 유용한 분자를 결합하는 상호작용 모듈로서 기능하며, 구조의 변화는 주로 특정 단백질의 결합 특성을 변화시키는 역할을 한다.
아연 집게는 인간 게놈의 유전자의 3%에서 발견될 수 있다. 게다가 아연 집게는 다양한 치료와 연구 능력에 매우 유용하게 쓰인다. 특정 염기서열에 대한 친화력을 가지는 아연 집게는 활성 연구의 영역이며, 아연 집게 핵산 분해 효소와 아연 집게 전사인자는 현재까지 실현되어야 할 가장 중요한 두 가지 응용 분야이다.
도메인
[편집]아연 집게 도메인(Znf)은 표적 분자와 직렬 접촉하는 집게 모양의 돌출부가 여러개 들어 있는 비교적 작은 단백질 모티브다. 이러한 도메인들 중 일부는 아연과 결합하지만, 많은 도메인들은 철과 같은 다른 금속을 결합하거나 전혀 결합하지 않는다. 예를 들어, 몇몇 족들은 염다리를 형성하여 집게와 같은 주름을 안정시킨다. 그들은 처음에 아프리카발톱개구리의 전사 인자 TFIIIA에서 DNA 결합 모티브로 확인되었으나, 현재는 DNA, RNA, 단백질, 지질들을 결합하는 것으로 인식되고 있다.[4][5][6][7][8] 이들의 결합 특성은 집게 영역의 아미노산 순서와 집게 사이의 연결점, 그리고 고차 구조와 집게의 수에 따라 달라진다. 아연 집게 도메인은 집게가 서로 다른 결합 특성을 가질 수 있는 클러스터에서 흔히 발견된다. 아연 집게 모티브는 몇 가지 관련이 없는 단백질 수퍼 패밀리에서 발생하며, 순서와 구조가 모두 다르다. 이들은 같은 부류의 구성원들(예 : 일부 결합 DNA, 다른 단백질) 사이의 결합에서 상당한 다기능성을 보여 아연 집게 모티브가 전문화된 기능을 발전시킨 안정적임을 시사한다. 예를 들어 아연 집게 단백질의 기능은 유전자 전사, 번역, 전령 RNA 트래피킹, 세포 골격 조직, 상피 발달, 세포 부착, 단백질 접힘, 크로마틴 리모델링, 아연 감지 등에서 사용된다.[9] 아연 결합 모티브는 안정된 구조로, 표적을 묶을 때 순응적인 변화를 거의 겪지 않는다.
계층
[편집]처음에는 아연 집게라는 용어가 아프리카발톱개구리에서 발견된 DNA 결합 모티브를 설명하는 데만 사용되었지만, 현재는 아연 이온의 조정에 의해 관련된 어떤 수의 구조물을 가리키는 데 사용되고 있다. 일반적으로 아연 집게는 아연 이온을 시스테인과 히스티딘 잔기의 조합으로 배위 결합한다. 원래 이러한 잔기의 수와 순서는 다른 종류의 아연 집게(예 : Cys2His2, Cys4, Cys6)를 분류하는 데 사용되었다. 최근에는 아연 집게 단백질을 대신 분류하는 더 체계적인 방법이 사용되고 있다. 이 방법은 아연 집게 단백질을 접힌 영역의 단백질 골격의 전체적인 모양을 바탕으로 접힘 그룹으로 분류한다. 가장 흔한 아연 집게의 접힘 그룹은 Cys2His2, treble clef, 아연 리본이다.
다음 표는[10] 아연 집게의 다양한 구조와 주요 특징을 보여준다.
| 접힘 그룹 | 대표 구조 | 리간드 배치 |
|---|---|---|
| Cys2His2 | 
|
너클로부터의 2개의 리간드 및 나선의 C 말단으로부터의 2개의 리간드 |
| Gag knuckle | 
|
너클로부터의 2개의 리간드 및 짧은 나선 또는 루프로부터의 2개의 리간드 |
| Treble clef | 너클에서 나온 2개의 리간드와 나선의 N 말단에서 나오는 2개의 리간드 | |
| 아연 리본 | 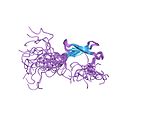
|
2개의 너클로부터 각각 2개의 리간드 |
| Zn2/Cys6 | 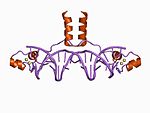
|
나선의 N 말단으로부터의 2개의 리간드 및 루프로부터의 2개의 리간드 |
| TAZ2 domain like | 두 나선의 말단에서 두 리간드 |
Cys2His2
[편집]| 아연 집게 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 상징 | ? | ||||||||
| Pfam | PF00096 | ||||||||
| Pfam clan | CL0361 | ||||||||
| InterPro | IPR007087 | ||||||||
| PROSITE | PS00028 | ||||||||
| |||||||||
Cys2His2 유사 접힘 그룹(C2H2)은 지금까지 가장 잘 알려진 아연 집게 계층이며 포유류에서 전사인자로 나타난다. 이러한 도메인은 간단한 ββα 접힘을 채택하고 아미노산 서열 모티프를 갖는다.[11]
- X2 - Cys - X2,4 - Cys - X12 - His - X3,4,5 - His
이러한 종류의 아연 집게는 RNA 결합 단백질 및 단백질-단백질 상호 작용을 매개하는 것과 같은 다양한 기능을 가질 수 있지만, Zif268(Egr1)과 같은 서열 특이적 DNA 결합 단백질에서의 역할로 가장 잘 알려져 있다. 이러한 단백질에서 개별 아연 집게 도메인은 전형적으로 단백질의 DNA 결합 도메인을 포함하는 2~3개, 또는 그 이상의 집게와 직렬 반복으로 발생한다. 이러한 직렬 배열은 DNA의 Major Groove에 결합 할 수 있으며 일반적으로 3bp 간격으로 떨어져 있다. 각 도메인의 알파 나선(인식 나선이라고도 함)은 DNA 염기에 서열 특이적 접촉을 할 수 있다. 단일 인식 나선으로부터의 잔기는 4개 이상의 염기와 접촉하여 인접한 아연 집게와의 접촉 패턴의 중첩 패턴을 생성 할 수 있다.
Gag-knuckle
[편집]| 아연 집게 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 상징 | ? | ||||||||
| Pfam | PF00098 | ||||||||
| InterPro | IPR001878 | ||||||||
| SMART | SM00343 | ||||||||
| PROSITE | PS50158 | ||||||||
| |||||||||
이 접힘 그룹은 회전(아연 너클)과 짧은 나선 또는 루프로 연결된 두 개의 짧은베타 가닥으로 정의되며 나선의 대부분과 베타 헤어핀 구조가 잘린 것이 고전적인 Cys2His2과 유사하다.
HIV 및 다른 레트로바이러스로부터의 레트로바이러스 뉴클레오캡시드(NC) 단백질은 이러한 모티프를 갖는 단백질의 예이다. HIV NC 단백질의 Gag-knuckle은 아연 집게 억제제로 알려진 약물 종류의 표적이다.
Treble-clef
[편집]Treble-clef 모티프는 길이와 형태가 다른 루프와 두 번째 베타 헤어핀 구조가 아연 결합에 대해 두 개의 리간드를 각각 기여하는 N 말단의 베타 헤어핀 구조와 C 말단의 알파 나선 구조로 구성된다. N 말단 베타 헤어핀 구조와 C 말단 알파 나선 구조 사이에 존재한다. 종종 이들 집게는 서로 서열 또는 기능적 유사성을 공유하지 않는 다양한 단백질 그룹에 존재한다. Treble-clef 아연 집게를 함유한 가장 잘 알려진 단백질은 핵 수용체이다.
아연 리본
[편집]| 아연 집게 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 상징 | ? | ||||||||
| Pfam | PF08271 | ||||||||
| Pfam clan | Zn_Beta_Ribbon | ||||||||
| InterPro | IPR013137 | ||||||||
| PROSITE | PS51134 | ||||||||
| |||||||||
아연 리본 접힘은 2개의 구조적으로 유사한 아연 결합 서브 사이트를 형성하는 2개의 베타 헤어핀 구조를 특징으로 한다.
Zn2/Cys6
[편집]| Fungal Zn(2)-Cys(6) binuclear cluster domain | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 상징 | Zn_clus | ||||||||
| Pfam | PF00172 | ||||||||
| InterPro | IPR001138 | ||||||||
| SMART | GAL4 | ||||||||
| PROSITE | PS00463 | ||||||||
| CDD | cd00067 | ||||||||
| |||||||||
이 부류의 표준 구성원은 2개의 아연 이온이 6개의 시스테인 잔기에 의해 결합된 이핵성 아연 클러스터를 함유한다. 이 아연 집게는 효모 Gal4 단백질을 포함한 여러 전사인자에서 찾을 수 있다.
| 아연 집게 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 상징 | ? | ||||||||
| Pfam | PF01530 | ||||||||
| InterPro | IPR002515 | ||||||||
| |||||||||
| 아연 집게 | |
|---|---|
| 식별자 | |
| 상징 | ? |
Miscellaneous
[편집]| 아연 집게 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 상징 | ? | ||||||||
| Pfam | PF01530 | ||||||||
| InterPro | IPR002515 | ||||||||
| |||||||||
| 아연 집게 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 상징 | ? | ||||||||
| Pfam | PF06221 | ||||||||
| InterPro | IPR009349 | ||||||||
| |||||||||
응용
[편집]다양한 단백질 공학 기법을 사용하여 아연 집게의 DNA 결합 특성과 그러한 공학적 아연 집게의 직렬 반복을 변경하여 원하는 게놈 DNA 염기서열을 대상으로 할 수 있다.[11] 전사 활성자나 억제자와 같은 두 번째 단백질 영역을 주어진 유전자의 촉진유전자 근처에 결합하는 일련의 공학적 아연 집게에 결합하는 것은 그 유전자의 전사를 바꾸는 데 사용될 수 있다. DNA를 갈라놓거나 수정하는 아연 집게 배열과 단백질 도메인 사이의 융합은 원하는 게놈 위치의 활동들을 목표로 하는 데 사용될 수 있다. 공학적 아연 집게 배열의 가장 일반적인 응용법은 아연 집게 전사인자와 아연 집게 핵산 분해 효소이지만 다른 응용법도 개발되었다. 대표적인 공학적 아연 집게 배열은 3개에서 6개의 개별 아연 집게 모티브를 가지고 있으며 길이가 9개에서 18개의 염기쌍에 이르는 결합 대상 부분을 가지고 있다. 6개의 아연 집게 모티브가 있는 배열은 포유류 게놈에서 특이할 가능성이 충분히 있는 대상을 묶기 때문에 특히 매력적이다.[12]
아연 집게 핵산 분해 효소
[편집]공학적 아연 집게 배열은 종종 아연 집게 핵산 분해 효소를 생성하기 위해 DNA 절단 도메인(일반적으로 FokI의 갈라짐 영역)에 융합된다. 이러한 아연 집게 융합체는 드로소필라 예쁜꼬마선충, 노랑초파리, 제브라 다니오[13], 담배, 옥수수[14], 얼룩말, 다양한 종류의 포유류 세포[15], 쥐를 포함한 많은 고등 유기체의 게놈을 조작하는 데 유용한 시약이 되었다.[16] 원하는 게놈 위치에 대한 이중 가닥 절단을 대상으로 하는 것은 비상동성 DNA 수선 경로의 오류 발생 가능성이 높은 성질 때문에 프레임 이동 돌연변이를 유전자의 암호화 순서에 도입하는 데 사용될 수 있다. 만약 상동성 DNA 공여자 서열을 사용한다면, 유전자좌는 상동성 지정 수선 경로를 통해 정의된 서열로 변환될 수 있다. 진행 중인 임상 실험은 CD4+ 인간 T세포의 CCR5 유전자를 교란시키는 아연 집게 핵산 분해 효소를 후천성면역결핍증후군의 잠재적 치료제로 평가하고 있다.[17]
아연 집게 배열의 공학적 방법
[편집]비록 몇몇 집단은 인간 전사인자 SP1에 기초한 아연 집게 배열을 사용했지만, 대부분의 공학적 아연 집게 배열은 높은 친화력으로 9bp 배열을 집합적으로 묶는 세 개의 개별 아연 집게 모티브를 가지고 있다.[18] DNA에 결합된 이 단백질의 구조는 1991년에 발견되었고[19], 공학적 아연 집게 배열에 대한 많은 연구를 했다. 1994년과 1995년에, 많은 집단이 Zif268의 아연 집게의 특이성을 바꾸기 위해 마커를 사용했다.[20][21][22][23] 현재 공학적 아연 집게 배열, 모듈식 조립 및 세균 선택 시스템을 생성하는 데 사용되는 두 가지 주요 방법이 있으며, 어떤 방법이 대부분의 용도에 가장 적합한지에 대한 논란이 있다.[24][25]
예시
[편집]이 항목은 진핵생물에서 발견된 CysCysHisCys(C2HC) 유형 아연 집게 도메인을 나타낸다. 이러한 도메인을 포함하는 단백질은 다음과 같다.
같이 보기
[편집]참고
[편집]각주
[편집]- ↑ “Zinc fingers: a novel protein fold for nucleic acid recognition”. 《Cold Spring Harbor Symposia on Quantitative Biology》 52: 473–82. 1987. doi:10.1101/sqb.1987.052.01.054. PMID 3135979.
- ↑ “Xenopus transcription factor A requires zinc for binding to the 5 S RNA gene”. 《The Journal of Biological Chemistry》 258 (23): 14120–5. December 1983. PMID 6196359.
- ↑ “Zinc fingers and other metal-binding domains. Elements for interactions between macromolecules”. 《The Journal of Biological Chemistry》 265 (12): 6513–6. April 1990. PMID 2108957.
- ↑ “Zinc finger peptides for the regulation of gene expression”. 《Journal of Molecular Biology》 293 (2): 215–8. October 1999. doi:10.1006/jmbi.1999.3007. PMID 10529348.
- ↑ “Multiple modes of RNA recognition by zinc finger proteins”. 《Current Opinion in Structural Biology》 15 (3): 367–73. June 2005. doi:10.1016/j.sbi.2005.04.004. PMID 15963892.
- ↑ “Zinc finger proteins: getting a grip on RNA”. 《Current Opinion in Structural Biology》 15 (1): 94–8. February 2005. doi:10.1016/j.sbi.2005.01.006. PMID 15718139.
- ↑ “Sticky fingers: zinc-fingers as protein-recognition motifs”. 《Trends in Biochemical Sciences》 32 (2): 63–70. February 2007. doi:10.1016/j.tibs.2006.12.007. PMID 17210253.
- ↑ “Zinc fingers--folds for many occasions”. 《IUBMB Life》 54 (6): 351–5. December 2002. doi:10.1080/15216540216035. PMID 12665246.
- ↑ “Zinc finger proteins: new insights into structural and functional diversity”. 《Current Opinion in Structural Biology》 11 (1): 39–46. February 2001. doi:10.1016/S0959-440X(00)00167-6. PMID 11179890.
- ↑ “Structural classification of zinc fingers: survey and summary”. 《Nucleic Acids Research》 31 (2): 532–50. January 2003. doi:10.1093/nar/gkg161. PMC 140525. PMID 12527760.
- ↑ 가 나 “Design and selection of novel Cys2His2 zinc finger proteins”. 《Annual Review of Biochemistry》 70: 313–40. 2001. doi:10.1146/annurev.biochem.70.1.313. PMID 11395410.
- ↑ “Design of polydactyl zinc-finger proteins for unique addressing within complex genomes”. 《Proceedings of the National Academy of Sciences of the United States of America》 94 (11): 5525–30. May 1997. Bibcode:1997PNAS...94.5525L. doi:10.1073/pnas.94.11.5525. PMC 20811. PMID 9159105.
- ↑ “[3H]MK801 binding to the N-methyl-D-aspartate receptor reveals drug interactions with the zinc and magnesium binding sites”. 《The Journal of Pharmacology and Experimental Therapeutics》 247 (3): 1025–31. December 1988. PMID 2849655.
- ↑ “Precise genome modification in the crop species Zea mays using zinc-finger nucleases”. 《Nature》 459 (7245): 437–41. May 2009. Bibcode:2009Natur.459..437S. doi:10.1038/nature07992. PMID 19404259.
- ↑ “Progress and prospects: zinc-finger nucleases as gene therapy agents”. 《Gene Therapy》 15 (22): 1463–8. November 2008. doi:10.1038/gt.2008.145. PMC 2747807. PMID 18784746.
- ↑ “Knockout rats via embryo microinjection of zinc-finger nucleases”. 《Science》 325 (5939): 433. July 2009. Bibcode:2009Sci...325..433G. doi:10.1126/science.1172447. PMC 2831805. PMID 19628861.
- ↑ Tebas P, Stein D (2009). “Autologous T-Cells Genetically Modified at the CCR5 Gene by Zinc Finger Nucleases SB-728 for HIV”. ClinicalTrials.gov.
- ↑ “DNA binding site of the growth factor-inducible protein Zif268”. 《Proceedings of the National Academy of Sciences of the United States of America》 86 (22): 8737–41. November 1989. Bibcode:1989PNAS...86.8737C. doi:10.1073/pnas.86.22.8737. PMC 298363. PMID 2510170.
- ↑ “Zinc finger-DNA recognition: crystal structure of a Zif268-DNA complex at 2.1 A”. 《Science》 252 (5007): 809–17. May 1991. Bibcode:1991Sci...252..809P. doi:10.1126/science.2028256. PMID 2028256.
- ↑ “Zinc finger phage: affinity selection of fingers with new DNA-binding specificities”. 《Science》 263 (5147): 671–3. February 1994. Bibcode:1994Sci...263..671R. doi:10.1126/science.8303274. PMID 8303274.
- ↑ “In vitro selection of zinc fingers with altered DNA-binding specificity”. 《Biochemistry》 33 (19): 5689–95. May 1994. doi:10.1021/bi00185a004. PMID 8180194.
- ↑ “Toward a code for the interactions of zinc fingers with DNA: selection of randomized fingers displayed on phage”. 《Proceedings of the National Academy of Sciences of the United States of America》 91 (23): 11163–7. November 1994. Bibcode:1994PNAS...9111163C. doi:10.1073/pnas.91.23.11163. PMC 45187. PMID 7972027.
- ↑ “Building zinc fingers by selection: toward a therapeutic application”. 《Proceedings of the National Academy of Sciences of the United States of America》 92 (2): 344–8. January 1995. Bibcode:1995PNAS...92..344W. doi:10.1073/pnas.92.2.344. PMC 42736. PMID 7831288.
- ↑ “Genome editing with modularly assembled zinc-finger nucleases”. 《Nature Methods》 7 (2): 91; author reply 91–2. February 2010. doi:10.1038/nmeth0210-91a. PMC 2987589. PMID 20111032.
- ↑ “Reply to "Genome editing with modularly assembled zinc-finger nucleases"”. 《Nat. Methods》 7 (2): 91–2. February 2010. doi:10.1038/nmeth0210-91b. PMC 2987589.
- ↑ “MYST family histone acetyltransferases in the protozoan parasite Toxoplasma gondii”. 《Eukaryotic Cell》 4 (12): 2057–65. December 2005. doi:10.1128/EC.4.12.2057-2065.2005. PMC 1317489. PMID 16339723.
- ↑ “The histone H4 acetyltransferase MOF uses a C2HC zinc finger for substrate recognition”. 《EMBO Reports》 2 (2): 113–8. February 2001. doi:10.1093/embo-reports/kve022. PMC 1083818. PMID 11258702.
- ↑ “Myelin transcription factor 1 (Myt1) of the oligodendrocyte lineage, along with a closely related CCHC zinc finger, is expressed in developing neurons in the mammalian central nervous system”. 《Journal of Neuroscience Research》 50 (2): 272–90. October 1997. doi:10.1002/(SICI)1097-4547(19971015)50:2<272::AID-JNR16>3.0.CO;2-A. PMID 9373037.
- ↑ “ST18 is a breast cancer tumor suppressor gene at human chromosome 8q11.2”. 《Oncogene》 23 (57): 9295–302. December 2004. doi:10.1038/sj.onc.1208131. PMID 15489893.
